“[…] la distinzione tra passato, presente e futuro è soltanto un’illusione, anche se ostinata”. Einstein
Una tappa cruciale
La coincidenza tra un crollo demografico e una fusione
cromosomica fa pensare a un evento di speciazione (Vedi sotto).
Nel settembre scorso veniva pubblicata su «Science» la ricerca con la quale abbiamo documentato una
drammatica strozzatura demografica intervenuta nella storia remota dei nostri antenati diretti, tra 930.000 e 813.000 anni fa. A seguito dell’analisi di dati genetici di popolazioni
umane attuali e di sofisticati calcoli bioinformatici, basati su un algoritmo innovativo elaborato dai nostri colleghi cinesi, ci risulta che
il bottleneck (È possibile che popolazioni solitamente numerose di tanto in tanto attraversino periodi difficili, nei quali sopravvive soltanto un piccolo numero di individui. Durante queste fasi di contrazione numerica della popolazione la deriva genetica può portare a una riduzione della variabilità genetica) si sia verificato ben prima della comparsa di Homo sapiens e che rappresenti l’origine del discusso
taxon (vedi sotto) Homo heidelbergensis, inteso come ultimo antenato comune dell'Homo Sapiens, dei
Neanderthal e dei «Denisova».
Nel clamore mediatico, era però passata
in secondo piano la concomitanza evolutiva
con quanto stimato l’anno prima da ricercatori dell’Istituto di informatica dell’Università di
Varsavia.
In un articolo su «BMC Genomics»,
Barbara Poszewiecka e colleghi avevano usato
un loro algoritmo, altrettanto innovativo, che aveva consentito di valutare quando sarebbe avvenuta la fusione del cromosoma 2 che Homo sapiens ha ereditato a seguito
di una mutazione cromosomica, che sarebbe
avvenuta durante il percorso evolutivo che ha portato alla nostra specie.
La loro stima coincide nei tempi con la nostra: circa
900.000 anni fa, con un intervallo di confidenza tra 400.000 e 1,5 milioni di anni fa.
Un cromosoma in meno
Come molti sanno, è una questione intrigante. Il cromosoma 2 è infatti il secondo
più grande cromosoma del genoma di Homo
sapiens. Ce ne sono due copie in ogni cellula, sono composti da circa 242 milioni di
nucleotidi, contengono oltre 1.600 geni e rappresentano l’8 per cento del nostro DNA; ma
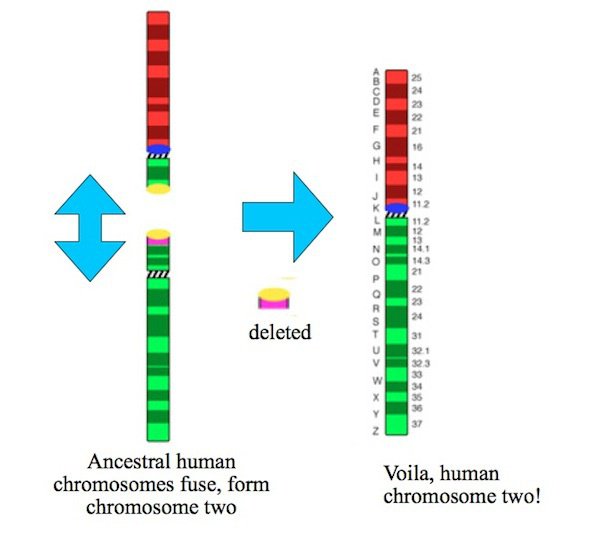
il punto che più ci interessa qui è che il cromosoma 2 sarebbe il risultato della fusione di
due cromosomi ancestrali.
Questa mutazione
è nota per la notevole omologia che si riscontra nel bandeggio tra il cromosoma umano
e i due (2p e 2q) presenti nel genoma dei nostri parenti più stretti fra i primati viventi, gli
scimpanzé; ma lo stesso è vero per altre scimmie antropomorfe.
Anche la presenza di centromeri (o «costrizione primaria») e telomeri
(segmenti terminali dei cromosomi), anomali
o vestigiali, è la chiara traccia di due cromosomi che negli scimpanzé sono rimasti separati mentre in noi sono fusi.
Lo dimostrarono
nel 1991 J.W. Ijdo e collaboratori sui «Proceedings of the National Academy of Sciences», e
altri in seguito.
Ne consegue che, mentre tutte le grandi scimmie antropomorfe hanno 24
coppie di cromosomi, noi umani ne abbiamo
solo 23 coppie.
Un evento cruciale, ma quando?
A prima vista, verrebbe da pensare che
questa fusione cromosomica sia avvenuta in
un momento cruciale della nostra evoluzione. Sembrerebbe dunque ragionevole immaginare che una così vistosa mutazione sia
intervenuta con la separazione evolutiva dagli antenati degli scimpanzé, cioè con i primi ominidi bipedi, circa 5-6 milioni di anni fa,
oppure all’alba del genere Homo, intorno a 2,5
milioni di anni fa.
Che invece questa mutazione sia alle origini dell’antenato comune fra l'Homo Sapiens,
i Neanderthal e i Denisova a me sembra sorprendente e oltremodo interessante.
Fra gli
altri aspetti, ci conferma che il bottleneck di
circa 900.000 anni fa sarebbe stato all’origine
di una vera e propria speciazione e che questo passaggio evolutivo, da forme precedenti del genere Homo (verosimilmente Homo ergaster) alla specie Homo heidelbergensis (o chi
per lei), sia stato anch’esso un evento cruciale
nella nostra storia evolutiva.
di Giorgio Manzi
LA SPECIAZIONE
La speciazione è un processo evolutivo grazie al quale si formano nuove specie da quelle preesistenti.
Il concetto di speciazione è stato essenzialmente sviluppato da Ernst Mayr, Julian Huxley e Teodosij Dobžanskij.
La speciazione risulta dalla selezione naturale e/o dalla deriva genetica, che sono i due motori dell'evoluzione. Questo fenomeno è una delle colonne portanti del neodarwinismo.
Modalità di speciazione
Le diverse modalità di speciazione possono essere classificate in due ampie categorie: la divergenza adattiva, che consiste nello sviluppo graduale e in tempi lunghi dell'isolamento riproduttivo, sotto la spinta della deriva genetica o della selezione naturale, e la speciazione improvvisa, che avviene bruscamente e riguarda in massima parte la nascita di individui (di solito vegetali) con corredo cromosomico poliploide (es. tabacco, cotone, patata).
Divergenza adattiva
Vi sono quattro differenti modalità di speciazione per divergenza adattiva: allopatrica, parapatrica, peripatrica e simpatrica, associate al livello di isolamento geografico delle popolazioni speciantesi.
Speciazione allopatrica
Esistono due grandi tipi di speciazione allopatrica, entrambi i modelli proposti da Ernst Mayr:
Speciazione dicopatrica o per vicarianza
Speciazione peripatrica o per dispersione
La speciazione allopatrica prevede l'esistenza di barriere geografiche che separando due popolazioni di individui della stessa specie in due territori differenti (definiti isole), interrompono il flusso genico della popolazione iniziale portando alla diversificazione di due specie differenti (vicarianza).
L'isolamento geografico può essere legato alla presenza di barriere naturali preesistenti quali montagne, deserti, mari, etc. oppure essere il risultato di una modificazione ambientale (innalzamento del livello del mare, deviazione di un corso d'acqua, costruzione di barriere artificiali, etc.).
Quando due popolazioni di individui della stessa specie si trovano in condizioni di isolamento, le nuove condizioni ambientali favoriscono per selezione ulteriori cambiamenti genetici, per cui, se l'isolamento persiste per un periodo sufficiente, la neo-specie non sarà più in grado di incrociarsi con la popolazione di origine.
L'esempio classico, da cui prende origine questa teoria, è quello dei "fringuelli di Darwin", osservati da Charles Darwin sulle isole Galápagos.
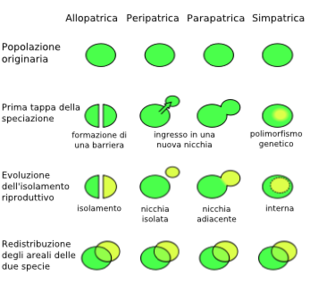
Differenti modalità di speciazione
Speciazione parapatrica
Si parla di speciazione parapatrica quando la divergenza avviene all'interno di popolazioni che non sono totalmente isolate geograficamente ma possiedono una ristretta zona di contatto. Le migrazioni tra popolazioni sono tuttavia limitate poiché queste ultime si perpetuano all'interno di condizioni ambientali differenti (per esempio gradienti climatici). La selezione naturale ha dunque un ruolo importante in questa modalità di speciazione.
Se le due specie hanno acquisito completo isolamento riproduttivo possono sovrapporsi, in base alle preferenza di habitat.
Se si sono sviluppate barriere di isolamento riproduttivo ma non compatibilità ecologica, gli areali si mantengono parapatrici.
Se non sono state acquisite barriere anti-ibridazione, si forma una zona di contatto che porta alla formazione di ibridi, nei quali possono evolvere barriere post-copula.
Speciazione peripatrica
La speciazione peripatrica, o speciazione per "effetto del fondatore" avviene quando un piccolo numero di individui costituisce una nuova popolazione ai margini dell'areale della specie di origine, ad esempio colonizzando una piccola isola vicina alla costa. La nuova popolazione può rapidamente evolvere in una nuova specie. A questo modello sono riferibili i casi di semi-specie, circoli di specie, super specie.
Speciazione simpatrica
La speciazione simpatrica avviene quando due popolazioni non isolate geograficamente si evolvono in specie distinte grazie alla presenza di polimorfismo nel tempo. In questo caso la selezione naturale gioca un ruolo cruciale nella divergenza delle popolazioni. Il fenomeno presenta tuttora degli aspetti controversi ma è stato ben documentato in alcuni casi, per esempio nei pesci di acqua dolce della famiglia dei Ciclidi.
I principali studi in questo campo riguardano gli esperimenti sulla Drosophila.
La speciazione simpatrica può avvenire per formazione di una criptospecie, ossia di una popolazione non più interfeconda con la specie di origine, a causa di alterazioni del cariotipo.
Questo meccanismo può rientrare tra le modalità di speciazione improvvisa.
La speciazione simpatrica si può vedere in paleontologia quando specie e generi, indagati con criterio stratigrafico, dimostrano le derivazioni fra loro e i rapporti filetici; tali taxa si susseguono nel tempo geologico, nell'ambito di un dominio paleogeografico isolato. Vedi l'esempio degli ammoniti Hildoceratidae in Appennino durante il Toarciano della Tetide occidentale (Giurassico inferiore; Venturi et al., 2010).
Speciazione improvvisa
La speciazione improvvisa, o istantanea, è un processo che riguarda prevalentemente gli organismi vegetali e si verifica in tempi brevi. Tipica causa è il fenomeno della poliploidia, che consiste in un aumento del numero di cromosomi rispetto al normale assetto diploide.
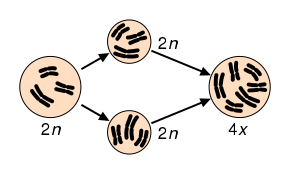
Meccanismo della poliploidia
Meccanismo della poliploidia
La poliploidia può avvenire per:
- una non-disgiunzione dei cromosomi durante il processo mitotico o meiotico;
- un processo meiotico o mitotico avvenuto senza errori, ma senza la successiva citodieresi.
Dunque, prendendo in esame un qualunque individuo diploide, si ha una situazione del genere:
- l'individuo effettua la meiosi;
- durante la meiosi, avviene una non-disgiunzione dei cromosomi: i gameti risulteranno quindi diploidi;
- i gameti si incrociano tra loro: lo zigote risultante sarà tetraploide, ossia avrà un assetto cromosomico doppio rispetto al suo ascendente originario, cioè l'organismo iniziale preso in considerazione.
Tuttavia, la formazione di criptospecie avviene perlopiù a livello di ibridi, cioè di individui frutto dell'incrocio di due specie differenti e quindi sterili. Nel caso degli organismi vegetali, gli ibridi sterili sopperiscono attraverso la riproduzione
Nel 2011 è stato documentato il caso di speciazione simpatrica in organismi termofili.
TAXON
Un taxon (plurale taxa, dal greco taxis, "ordinamento") o unità tassonomica, è un raggruppamento di oggetti o organismi, distinguibili morfologicamente dagli altri per una caratteristica comune e che possono più precisamente anche essere organizzati attraverso la sistematica in una gerarchia, dando inizio ad una classificazione scientifica. La scienza che definisce i taxa si chiama tassonomia.
A ogni gruppo viene assegnato un nome in latino, una descrizione e un "tipo", a seconda se il taxón è una specie, un campione oppure un esemplare concreto. Ogni descrizione formale di taxón viene associata al nome dell'autore o autori che l'hanno realizzata, i quali figureranno dopo il nome.
Lo scopo di classificare gli organismi in taxa formalmente definiti, invece che tramite gruppi informali, è quello di fornire gruppi la cui circoscrizione (cioè, quali organismi li compongono) resti in un ambito ristretto e la cui denominazione abbia valore universale, indipendentemente dalla lingua utilizzata per la comunicazione. Si può notare che i taxa esistono dentro una classificazione data, soggetta a mutamenti e sopra la quale possono presentarsi discrepanze e, rispetto a certe denominazioni problematiche, si è obbligati a specificare di quale autore è il nome che si sta utilizzando.
In ordine gerarchico, secondo lo schema di classificazione linneana, diamo la seguente lista generale (ordinata dal generale al particolare) delle categorie tassonomiche alle quali vengono associati i diversi taxa.
Regno
Phylum (animali o piante) o Divisione (piante)
Classe
Ordine
Famiglia
Genere
Specie
(Filastrocca: Rex Fedele Cane Obbediente Fa Gesti Studiati)
CLASSIFICAZIONE COMPLETA DELL'UOMO
Uomo (Homo sapiens)
Dominio Eukaryota
Regno Animalia
Phylum Chordata
Subphylum Vertebrata
Superclasse Tetrapoda
Classe Mammalia
Sottoclasse Placentati
Infraclasse Eutheria
Ordine Primates
Sottordine Haplorrhini
Superfamiglia Hominoidea
Famiglia Hominidae
Sottofamiglia Homininae
Tribù Hominini
Sottotribù Hominina
Genere Homo
Specie H. sapiens
CROMOSOMA 2
Con il nome di cromosoma 2 si indica, per convenzione, il secondo più grande cromosoma umano. Le persone presentano solitamente due copie del cromosoma 2, come di ogni autosoma. Il cromosoma 2 possiede all'incirca 242 milioni di nucleotidi. I due cromosomi 2 rappresentano all'incirca l'8% del DNA totale nelle cellule umane.
Il cromosoma 2 contiene di certo oltre 1.600 geni, ma si ritiene possa contenerne fino a 1.800.
Il numero di polimorfismi a singolo nucleotide (SNP) individuati è di quasi 900.000.
Si ritiene che questo cromosoma sia il risultato della fusione di due cromosomi ancestrali. La prova di questo starebbe nell'alta omologia tra il cromosoma umano e due cromosomi dello scimpanzé. Ad avvalorare questa ipotesi ci sarebbe anche la presenza di centromeri e telomeri vestigiali sul cromosoma umano, resti proprio dei due cromosomi presenti oggi nello scimpanzé.
I NUCLEOTIDI
In chimica, i nucleotidi sono unità ripetitive costitutive degli acidi nucleici (DNA e RNA).
Sono costituiti da tre gruppi:
- una base azotata, purinica o pirimidinica;
- uno zucchero a cinque atomi di carbonio (zucchero pentoso), che insieme alla base azotata costituisce un nucleoside;
- uno o più gruppi fosfato (residuo fosforico) che, insieme al nucleoside, completa il nucleotide.
Le basi puriniche sono adenina e guanina, nel DNA come nell'RNA; le basi pirimidiniche sono citosina e timina nel DNA, citosina e uracile nell'RNA.
Lo zucchero pentoso è il ribosio nell'RNA ed il desossiribosio nel DNA.
La presenza del residuo fosforico conferisce un carattere fortemente acido ai nucleotidi; per questo i loro nomi alternativi sono acido adenilico, acido guanilico, acido citidilico e acido uridilico. Nel DNA i nucleotidi (desossinucleotidi) prendono il nome di acido desossiadenilico, acido desossiguanilico, acido desossicitidilico e acido desossitimidilico.
L'aggiunta di uno o due altri residui fosforici nella catena produce i nucleosidi difosfato e trifosfato (NDP e NTP), fondamentali nel metabolismo energetico della cellula. I più importanti sono ADP e ATP, rispettivamente adenosina difosfato e adenosina trifosfato. Il passaggio da tri- a di- avviene tramite una reazione di idrolisi esoergonica con conseguente liberazione di energia che la cellula può utilizzare per le sue attività.
Nomenclatura
I nomi dei nucleotidi sono abbreviati in codici standard di tre o quattro lettere. Un'eventuale "d" iniziale sta per "desossi-", cioè indica che il nucleotide in questione è un desossiribonucleotide, come nel DNA; nel caso di un ribonucleotide semplicemente non è presente la "d", come nell'RNA. La seconda lettera indica il nucleoside, e precisamente:
- G sta per guanosina, corrispondente alla base azotata guanina
- A sta per adenosina, corrispondente alla base azotata adenina
- T sta per timidina (che non essendo presente nell'RNA, dove viene sostituita dall'uridina, è un desossinucleoside), corrispondente alla base azotata timina
- C sta per citidina, corrispondente alla base azotata citosina
- U sta per uridina (che non è presente nel DNA e prende il posto della timidina nell'RNA), corrispondente alla base azotata uracile
Desossicitidina 5'-monofosfato
La terza lettera indica la lunghezza della catena di gruppi fosfato attaccata al carbonio 5' dello zucchero. Nel DNA e nell'RNA c'è un solo gruppo fosfato per unità ripetitiva, quindi si utilizza solo il prefisso mono-, abbreviato in M; nella reazione di una DNA o di una RNA polimerasi, invece, ogni desossinucleotide in ingresso nella catena in costruzione è trifosfato, quindi nell'indicarlo si usa il prefisso tri-, abbreviato in T. La quarta lettera indica la presenza di gruppi fosfato (P).
Ad esempio, la desossicitidina 5'-monofosfato è abbreviata con dCMP; la d perché è un desossiribonucleotide, la C perché la base azotata contenuta è la citosina, "MP" perché lo zucchero che vi si trova (il desossiribosio) è monofosfato al carbonio 5'. (N.B. nella figura che segue sarebbe più corretto parlare di nucleotide monofosfato e nucleotide difosfato e non nucleoside monofosfato e nucleoside difosfato, in quanto è visibile il legame e la presenza della base azotata).
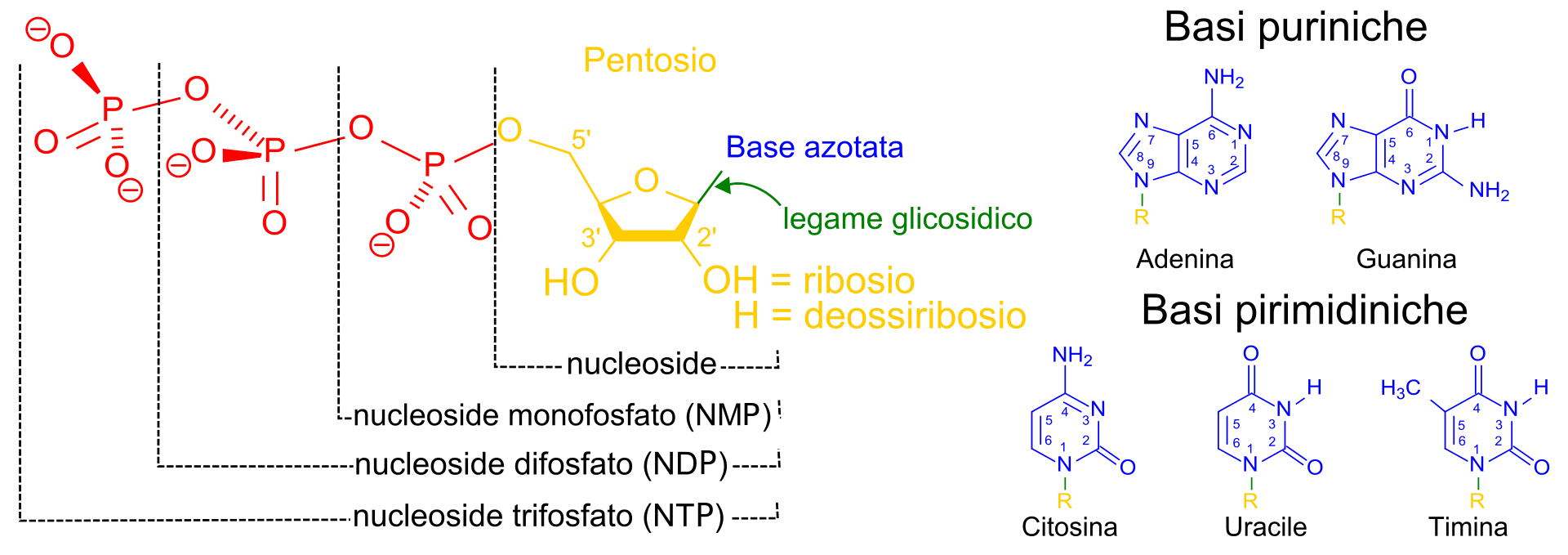
Nucleotide comune
Eugenio Caruso - 28 gennaio 2024

Tratto da le scienze